| |
| |
|
|
| |
| |
基因编辑后基因敲入检测试剂盒,简便·快速
Guide-it Knockin Screening Kit |
| |
Guide-it Knockin Screening Kit可高灵敏度地检测通过同源重组(homologous recombination, HR)所产生的基因编辑位点,可用于检测使用CRISPR/Cas9等技术进行基因编辑的批量或者单克隆细胞。该试剂盒采用了简单的基于荧光的检测方法,不论所敲入的插入物长度(从单碱基替换到更长的插入片段)或被编辑的基因组区域序列如何,都可以对由同源重组(HR)所产生的基因组编辑位点进行检测,这种检测方法简便可靠。
|
| 该试剂盒操作流程简便快速,主要包括基因组靶标位点PCR扩增,绿色和红色双色荧光检测的酶法测定。这个酶法测定实验,使用荧光读板机或定量PCR仪检测荧光信号即可进行测定,不需要其他特殊仪器。整个操作流程大约需要4个小时,由于荧光信号的检测与基因组靶标位点上目标序列的正确引入是正相关的关系,从而确保可以检测到突变。 |
| 在引入单核苷酸多态性(SNP)的应用中,该检测方法能够在混合和克隆细胞群中高灵敏度地检测出单核苷酸替换,可鉴定识别含有两个不同等位基因的杂合克隆(例如携带经编辑(SNP)和未经编辑(WT)各一个拷贝的等位基因)。对于敲入更长基因序列的应用,使用这种检测方法可以在插入序列的5'和3'末端,同时检测是否无缝插入。 |
| |
| ■ 特点 |
· 无需测序,快速确定是否发生了正确的基因敲入
· 可用于批量或者单克隆细胞检测
· 操作简单快速,4小时可完成检测
· 无需特殊仪器,使用荧光读板机或者定量PCR仪进行测定
· SNP分析理想选择,双色检测可同时检测两个不同等位基因(SNP和WT等)
|
| |
| ■ 应用 |
· 在经过编辑的细胞群中快速检测单核苷酸替换或准确敲入
· 在杂合克隆中同时检测已编辑和未编辑的等位基因
· 在进行单细胞克隆之前对杂合的细胞群进行编辑效率分析
|
| |
| ■ 在线工具 |
寡核苷酸DNA设计对于使用Guide-it Knockin Screening Kit进行SNP分析是非常必要的,这个在线工具可以让您轻松设计每个寡核苷酸DNA。
|
注意:此在线工具仅用于设计检测单碱基替换的寡核苷酸DNA,而不能用于检测长片段插入的寡核苷酸DNA的设计。
|
Oligo design tool for Guide-it SNP screening assays
|
| |
| ■ Tech Note |
在iPSC中引入与疾病相关的遗传变异,包括SNP,可以评估这些变异在相关细胞类型中的表型效应,并消除由于遗传背景引起的变异性,从而可以更精细地分析疾病机制并更快速地评估潜在疗法 (例如,药物筛选)。Guide-it Knockin Screening Kit提供了一种简便快速的方法,可以在繁琐耗时的单细胞克隆分离之前,从大量编辑后的细胞群和最终产生的克隆细胞群中,明确鉴定出成功的同源重组修复(homology-directed repair, HDR)。
|
详情点击图片查看
|
 |
| |
|
| ■ 使用实例 |
 |
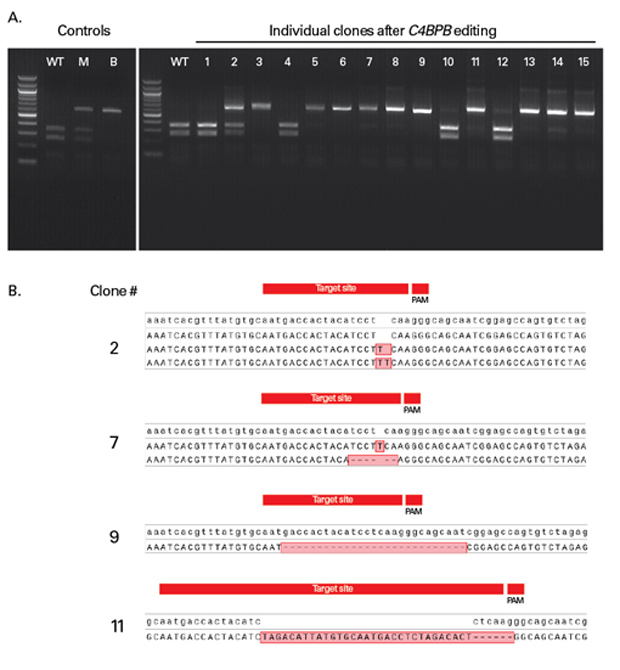
| 图1. 使用Guide-it Knockin Screening Kit检测敲入的插入片段全长 |
基因编辑后,通过FACS或有限稀释法分离的大量细胞群和克隆细胞系可能表现出多种模式,例如野生型(WT),引入插入/缺失(indel)或敲入了全长插入片段。从克隆细胞中提取DNA并扩增靶标位点,然后将PCR产物与两套不同的displacement oligo(紫色)和flap probe进行杂交:一个与插入片段的5'端杂交(Flap-probe oligo A; 绿色),另外一个与3'端杂交(Flap-probe oligo B; 橙色)。如果同源重组成功并且全长插入片段无缝正确插入的情况下,探针会与两端完全杂交,Guide-it Flapase剪切flap probes后产生绿色和红色荧光信号。当仅检测到一种荧光信号(红色或绿色)时,表明插入片段分别在5'端或3'端发生了缺失。如果没有检测到荧光,则表明靶标位点依然是野生型序列或者引入了indel。
|
| |
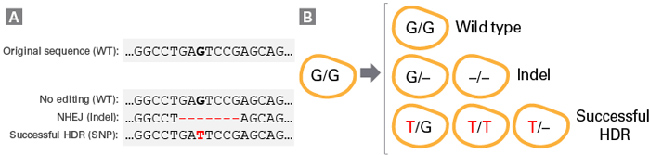 |
| 图2. 引入单碱基替换的常见结果 |
展示了使用基因编辑技术进行单碱基替换(G>T)的示例,此工作流程示例为G>A替换检测示例。
|
Panel A. 在基因组靶标位点的结果:当在靶标位点没有发生成功切割,或者成功切割后发生了基于非同源末端连接(NHEJ)的准确修复的情况下,靶标位点没有发生变化,结果仍然为野生型(WT)序列。当切割后发生了基于NHEJ的不准确修复的情况下,其结果是在靶标位点引入了插入或缺失(Indel),这可能会导致基因敲除(KO;极有可能的结果)。当切割后发生了准确的HDR时,则会在靶标位点引入SNP。
Panel B. 二倍体细胞中等位基因组合结果:在二倍体细胞中进行编辑时,每个等位基因的编辑结果可能会有所不同,从而产生多种可能的组合。如果没有发生基因编辑,细胞仍然可以保持纯合型(野生型;顶部);如果发生了不准确的NHEJ修复,会有一个或两个等位基因被修饰(Indel;中间);如果发生了成功的HDR,则可以获得一个或两个等位基因中引入了目标SNP的细胞(成功的HDR;底部)。
|
| |
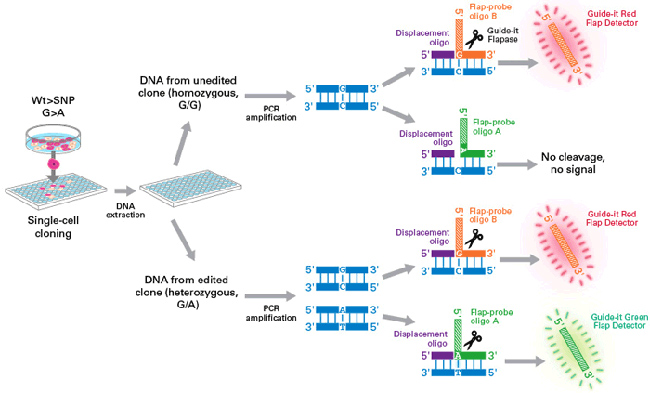 |
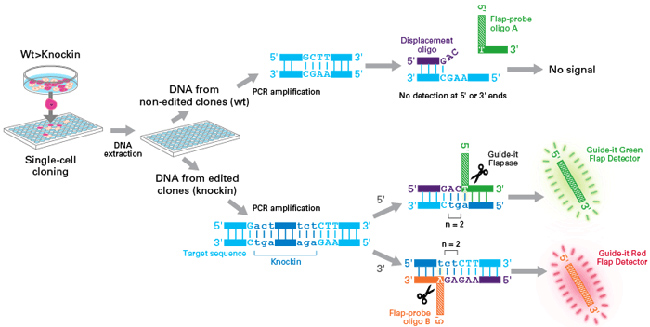
| 图3. 使用Guide-it Knockin Screening Kit进行SNP分析的流程 |
此示例工作流程演示了对G>A单碱基替换的分析,其中G是野生型碱基,被编辑为A。在基因编辑后,将单细胞扩培成为克隆细胞系,目标基因的靶标位点可能具有几种不同的基因型结果。扩增靶位点后,将PCR产物与不同的寡核苷酸探针进行杂交:displacement oligo(紫色)与flap-probe oligo A(绿色;编码SNP等位基因,A)或flap-probe oligo B(橙色;编码WT等位基因,G)组合使用。寡核苷酸退火至PCR产物后,Guide-it Flapase酶(剪刀所示)识别完全的碱基配对,并切割flap-probe oligo 5’端部分(绿色或橙色阴影)。切割下来的flaps由相对应的Guide-it flap detectors检测,并分别产生绿色或红色荧光信号。在上面的示例中,在靶标位点仅产生红色信号的克隆细胞系分析是纯合WT(G/G),而同时产生红色和绿色信号的则是携带已编辑和WT等位基因(G/A)的杂合克隆。
|
| |
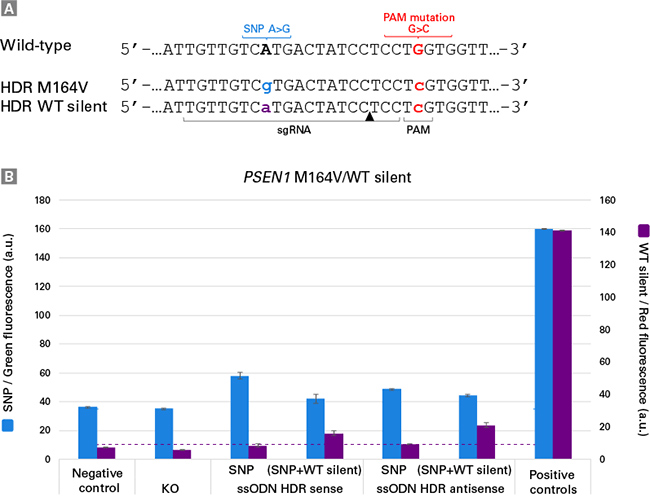 |
| 图4. 检测iPS细胞群中PSEN1位点的准确碱基替换 |
为了验证Guide-it Knockin Screening Kit的SNP检测功能,使用CRISPR/Cas9系统构建了编码PSEN1基因突变体A>G替换(M164V)的杂合型iPS细胞系,该突变体与早发型阿尔茨海默氏病相关。
|
Panel A. 使用包含PAM沉默突变的HDR模板,在PSEN1基因座上进行准确基因编辑:如果同源重组成功,编辑后的PSEN1基因座将编码邻近PAM序列发生沉默突变(红色,小写)的SNP(蓝色,小写)或者WT(紫色,小写)等位基因。
Panel B. 基因组编辑后在批量iPS细胞群中成功检测碱基替换:使用正义或者反义HDR模板进行基因编辑,然后使用Guide-it Knockin Screening Kit检测是否在每种情况下都成功发生了基因编辑。
设计displacement和flap-probe oligos,用于检测由相应HDR模板编码的邻近PAM序列发生沉默突变(G>C)的WT或者SNP等位基因,将分别产生红色和绿色荧光信号。在独立平行实验中,将Cas9蛋白质(阴性对照),Cas9-sgRNA RNP复合物(KO)或者RNP复合物和正义或者反义WT/SNP HDR模板混合物通过电穿孔的方式分别导入至细胞中。合成检测PAM序列沉默突变的WT或SNP编码序列的寡核苷酸探针,进行平行检测并作为阳性对照。对于每个电穿孔混合物中包含HDR模板的编辑实验组,可以在批量细胞群中检测到成功的HDR。阴性对照组没有显示绿色和红色荧光信号,这证实了flap-probe oligos的特异性,因为在未编辑的WT序列存在下它们没有产生假阳性荧光信号。 |
| |
| ■ 产品组份 |
| · MightyPrep Reagent for DNA |
| · Guide-it Knockin Positive Control Mix |
| · Guide-it Knockin Negative Control Mix |
| · Terra PCR Direct Polymerase Mix |
| · 2X Terra PCR Direct Buffer (with Mg2+, dNTP) |
| · Dilution Buffer |
| · RNase-free Water |
| · Annealing Buffer |
| · Flapase Buffer |
| · Guide-it Flapase |
| · Guide-it Green Flap Detector (40X) |
| · Guide-it Red Flap Detector (40X) |
|
| |
| ■ 保存 |
MightyPrep Reagent for DNA:4℃
|
其它:-20℃
|
| |
| |
产品详情请点击: |
| |
| |




.png)
.png)
.png)
.png)
.png)
.png)
.png)





.png)
.png)